13.12.2019 Neu entdeckter Schalter steuert Zellteilung bei Bakterien
Marburger Forschungsgruppe fand bislang unbekannten Mechanismus, der hilft, DNA auf Tochterzellen zu verteilen
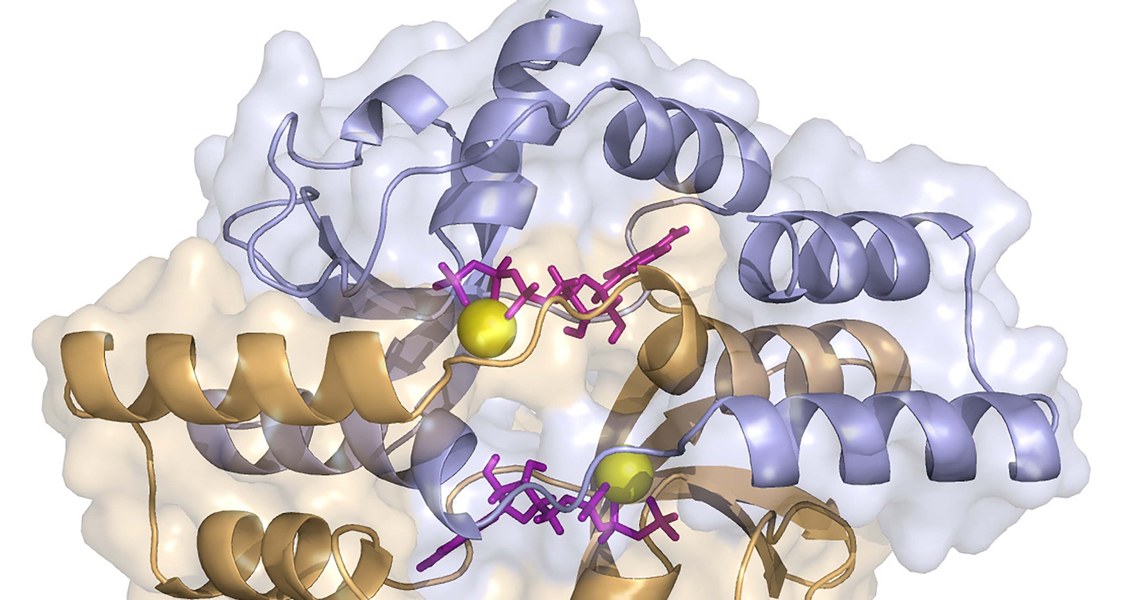
Ein neu entdeckter molekularer Mechanismus bereitet die Zellteilung von Bakterien vor, indem er die Verteilung des Erbguts auf die Tochterzellen beeinflusst. Das haben Wissenschaftlerinnen und Wissenschaftler der Philipps-Universität Marburg, des „LOEWE“-Zentrums für synthetische Mikrobiologie (SYNMIKRO) und des Max-Planck-Instituts für Terrestrische Mikrobiologie herausgefunden, die ihre Resultate in der Fachzeitschrift „Cell“ beschreiben.
Bevor sich Zellen teilen, verdoppeln sie ihr Erbgut; die beiden Kopien werden dann an die Tochterzellen verteilt. Was simpel klingt, erfordert eine reibungslos funktionierende Maschinerie, die aus zahlreichen Proteinen besteht. „Unsere Ergebnisse fügen dem gängigen Bild eine unvorhergesehene Ebene hinzu“, sagt der Marburger Mikrobiologe Professor Dr. Martin Thanbichler, Leitautor der neuen Studie. „Offenbar ist die bakterielle DNA-Verteilung komplexer als bislang angenommen.“
Viele Proteine wirken als Schalter, die wichtige Lebensprozesse in Zellen steuern – so auch die Verteilung des Erbguts. Damit die beiden DNA-Kopien zuverlässig zu ihren Zielorten verfrachtet werden, die in entgegengesetzten Richtungen liegen, müssen verschiedene Proteine ans Erbgut koppeln. Eine zentrale Komponente dieser Maschinerie haben die Forscherinnen und Forscher um Martin Thanbichler und den Marburger Biochemiker Professor Dr. Gert Bange nun genauer untersucht: Sie klärten die Struktur des Proteins ParB auf, analysierten das Verhalten seiner Abschnitte und überprüften die Wirkungsweise in lebenden Zellen.
Die Wissenschaftlerinnen und Wissenschaftler waren überrascht, als sich abzeichnete, dass ParB und seine Verwandten als molekulare Schalter wirken, die mit dem Nukleinsäure-Baustein CTP zusammenwirken – und nicht mit ATP oder GTP, wie sonst üblich. „Bindet CTP an einem bestimmten Proteinabschnitt, so schließen sich zwei ParB-Moleküle zu einer ringförmigen Struktur, welche sich um die Bakterien-DNA legt und so deren Verteilung ermöglicht“, erläutert Thanbichler. „Durch die anschließende Spaltung von CTP geht ParB dann in eine offene Form über, in der es die DNA später wieder verlassen kann.“
„ParB und seine Verwandten liegen bei vielen Bakterien in fast unveränderter Form vor“, legt Thanbichlers Mitarbeiter Manuel Osorio-Valeriano dar, der Erstautor des wissenschaftlichen Aufsatzes. „Unsere Daten zeigen, dass es sich dabei um eine gänzlich neue Proteinklasse handelt.“ Wie Thanbichler ergänzt, eröffnet diese Entdeckung „völlig neue Perspektiven für die Erforschung der Mechanismen, die dem lebenswichtigen Prozess der DNA-Verteilung bei der Zellteilung zugrunde liegen“.
Martin Thanbichler lehrt Mikrobiologie an der Philipps-Universität und ist daneben als Gruppenleiter am benachbarten Max-Planck-Institut für Terrestrische Mikrobiologie tätig. Professor Dr. Gert Bange hat eine Professur für Biochemie an der Philipps-Universität inne und ist stellvertretender Direktor des „LOEWE“-Zentrums für synthetische Mikrobiologie (SYNMIKRO). Das hessische Förderprogramm „LOEWE“, die Max-Planck-Gesellschaft und die Deutsche Forschungsgemeinschaft unterstützten die Forschungsarbeit finanziell.
Originalveröffentlichung: Manuel Osorio-Valeriano & al.: ParB-type DNA segregation proteins are CTP-dependent molecular switches, Cell 2019, doi: https://doi.org/10.1016/j.cell.2019.11.015
Weitere Informationen: Die dreidimensionale Gestalt eines ParB-Proteins zeichnet sich durch zwei Proteinketten aus sowie die daran gekoppelten CTP-Moleküle aus: Video