10.05.2022 Computerprogramm findet neue Zielorte für Medikamente
Größte Proteinfamilie enthält ungenutzte Angriffspunkte für Heilmittel

Eine computergestützte Analyse bereichert die Arzneimittelforschung: Ein internationales Team hat eine Software zu Hilfe genommen, um neue Angriffspunkte für künftige Medikamente zu finden. Die Wissenschaftlerinnen und Wissenschaftler entdeckten bei einer der größten Proteinfamilien mehrere neue Kopplungsstellen, an denen Wirkstoffe ansetzen können. Das Forschungsteam berichtet im Wissenschaftsmagazin „Nature Communications“ über die Ergebnisse.
Sie bilden die größte Familie von Proteinen, die als Ziel von Medikamenten dienen: G-Protein-gekoppelte Rezeptoren, kurz GPCR sind an zahlreichen Lebensvorgängen beteiligt. Hierzu zählen Entzündungen, die Verarbeitung von Sinnesempfindungen und die Wirkung von Hormonen. Das spiegelt sich auch in der Bedeutung dieser Proteine für die Medizin wider: Fast ein Drittel der rezeptpflichtigen Arzneimittel richtet sich gegen GPCR. „Um die Proteine zu blockieren oder zu stimulieren, benötigt man Wirkstoffe, die genau in eine der zahlreichen Bindungstaschen der Proteine passen“, erläutert der Pharmazeutische Chemiker Professor Dr. Peter Kolb von der Philipps-Universität Marburg, einer der Leitautoren des Fachaufsatzes.
GPCR-Proteine dienen dazu, Signale von der Außenseite einer Zelle in deren Inneres weiterzuleiten, so dass die Zelle auf Veränderungen in ihrer Umgebung reagieren kann. Um ihre Wirkung zu entfalten, koppeln Hormone und andere körpereigene Botenstoffe an die Bindungstaschen der GPCR-Proteine. Man kann die Wirkung dieser Botenstoffe unterdrücken, indem man die Bindungstaschen anderweitig besetzt.
„Die bisher genutzten Kopplungsstellen ähneln einander jedoch häufig sehr stark“, führt Kolb aus; „deswegen wirken Arzneistoffe oft zu wenig selektiv“. Dadurch steigt die Gefahr von Nebenwirkungen. So verwendet man Betablocker, um ein GPCR-Protein im Herzen zu blockieren; schaltet man dasselbe Ziel jedoch im Lungengewebe aus, kann dies Asthmaanfälle auslösen.
Um neue Bindungsstellen zu finden, taten sich die Arbeitsgruppen von Kolb und dessen Marburger Pharmazie-Kollegen Professor Dr. Moritz Bünemann zusammen, die beide dem Forschungsverbund GLUE angehören; GLUE steht für „GPCR Ligands for Underexplored Epitopes“. Das Konsortium geht der Frage nach, wie Medikamente maßgeschneidert werden können, so dass sie genau zu ihren Angriffspunkten passen, also weniger unbeabsichtigte Nebenwirkungen mit sich bringen. Das Land Hessen unterstützt das mittelhessische Projekt von 2020 bis 2023 mit insgesamt 4,6 Millionen Euro durch sein Förderprogramm „LOEWE“.
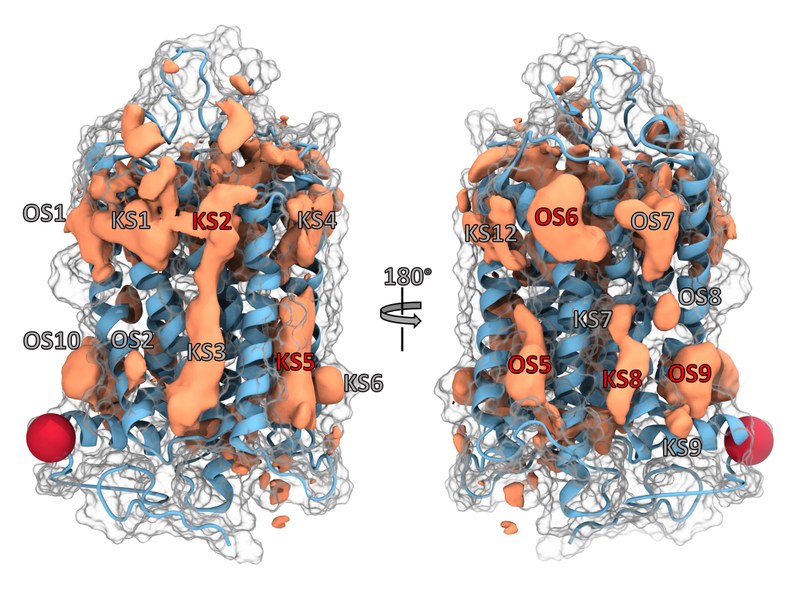
„Wir haben computergestützt nach alternativen Bindestellen auf 113 verschiedenen GPCR-Proteinen gesucht“, berichtet Kolbs Mitarbeiter und Koautor Janik B. Hedderich. Dabei simulierte und analysierte der Algorithmus, was passiert, wenn kleine Moleküle an unterschiedliche Stellen der Proteine koppeln. Die Studie deckt das gesamte Ensemble aller erkennbaren Taschen ab. „Auf diese Weise fanden wir tatsächlich mehrere Bindungstaschen, die bisher nicht als Ziel von Medikamenten dienen“, führt der Doktorand aus.
Experimentelle Untersuchungen an zwei der gefundenen Bindungstaschen ergänzen die Berechnungen. „Wir fügten Mutationen in diese zwei Taschen ein“, erläutert Mitverfasser Moritz Bünemann. „Die Ergebnisse bestätigen, dass diese Proteinabschnitte eine ausschlaggebende Funktion für die Aktivität der GPCR-Proteine innehaben.“
Die Studie stellt die derzeit umfassendste Analyse von GPCR-Bindungstaschen dar. „Unsere Ergebnisse sollen es leichter machen, Bindungspartner für die neu gefundenen Kopplungsstellen zu finden, um so die Aktivität von gesundheitsrelevanten GPCR-Proteinen zu steuern“, sagt Kolb.
Professor Dr. Peter Kolb ist Professor für Pharmazeutische Chemie an der Philipps-Universität. Janik Hedderich fertigt seine Doktorarbeit in Kolbs Arbeitsgruppe an. Professor Dr. Moritz Bünemann lehrt in Marburg Pharmakologie und Toxikologie. Er leitet das Institut für Pharmakologie und Klinische Pharmazie der Philipps-Universität und steht dem GLUE-Konsortium als Sprecher vor. Neben dem Marburger Team beteiligte sich eine kanadische Arbeitsgruppe an den Forschungsarbeiten.
Die Deutsche Forschungsgemeinschaft, das Horizon 2020-Programm der Europäischen Union, das GLUE-Projekt sowie die kanadische Förderorganisation CIHR unterstützten die beteiligten Wissenschaftlerinnen und Wissenschaftler finanziell.
Originalveröffentlichung: Janik B. Hedderich & al.: The pocketome of G protein-coupled receptors reveals previously untargeted allosteric sites, Nature Communication 2022, DOI: 10.1038/s41467-022-29609-6