20.12.2022 Forschungsteam klärt Wirkmechanismus eines Schlüsselenzyms auf
Internationale Forschungsgruppe studierte den Funktionsmechanismus einer Acetyltransferase
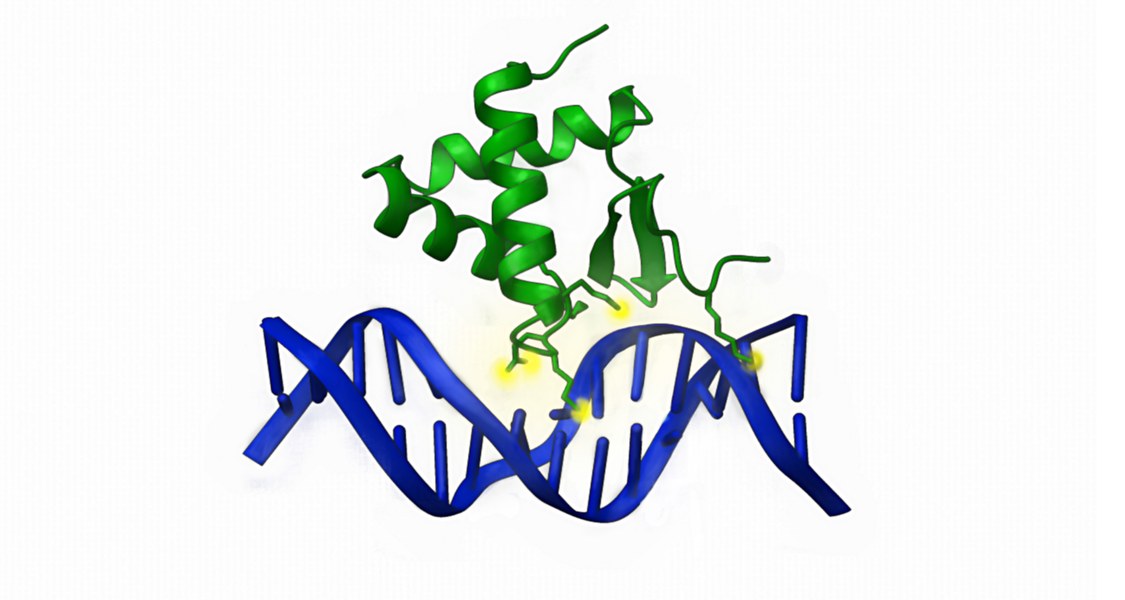
Eine internationale Forschungskooperation unter Marburger Leitung hat den Mechanismus aufgeklärt, mit dem das Enzym KAT6A zu seinen Zielgenen gebracht wird. Das Team weist nach, dass KAT6A mittels einer neu entdeckten Domäne an bestimmte Stellen der DNA andockt, an denen es die Verpackung der DNA lockert. Die Forschungsgruppe um den Biochemiker Dr. Robert Liefke berichtet im Fachblatt „Nucleic Acids Research“ über ihre Ergebnisse.
Die Erbsubstanz DNA liegt nicht nackt und bloß im Zellkern, sondern wird umhüllt von einer Schutzschicht aus Proteinen, dem so genannten Chromatin. Um einzelne Gene zu aktivieren, muss das Chromatin gelockert werden, so dass die DNA-Abschnitte zugänglich sind, auf denen das jeweilige Gen liegt. „KAT6A gehört zur Enzymklasse der Acetyltransferasen, die bei diesem Prozess entscheidend beteiligt sind“, sagt Robert Liefke, einer der Leitautoren der aktuellen Veröffentlichung.
Mutante Abwandlungen des Gens KAT6A kommen bei einer seltenen Erbkrankheit mit weltweit etwa 500 Patientinnen und Patienten vor, die unter anderem eine verzögerte Sprachentwicklung, geistige Behinderung, Komplikationen des Verdauungstraktes und Herzanomalien aufweisen. Auch an der Entstehung von akuter myeloischer Leukämie ist KAT6A beteiligt.
„Der genaue Mechanismus, wie KAT6A seine Zielgene findet und wie Mutationen von KAT6A zu Entwicklungsstörungen und Krebserkrankungen führen, war bislang nicht bekannt“, führt Liefke aus. Die Forschungsgruppe fand nun einen bisher unerforschten Abschnitt des KAT6A-Enzyms, das an bestimmte Stellen der DNA andockt, nämlich an so genannte CpG-Inseln. Dabei handelt es sich um Stellen, an denen sich die DNA-Bausteine C und G häufen. „CpG-Inseln bilden Schlüsselstellen für die Steuerung von Genen“, legt Liefke dar.
Die Vertreter der Acetyltransferasen sind dafür bekannt mit anderen Proteinen zusammenwirken, aber bisher wurde noch kein Beispiel beschrieben, welches direkt mit der DNA interagiert. „Nach unserem besten Wissen ist dies die erste Beschreibung eines Mitglieds dieser Enzymklasse mit einer spezifischen DNA-Bindefunktion.“
Die Wissenschaftlerinnen und Wissenschaftler überprüften experimentell, ob der von ihnen gefundene Enzymabschnitt tatsächlich dafür verantwortlich ist, dass KAT6A an die CpG-Inseln der DNA bindet. Zu diesem Zweck baute das Team Mutationen in die entsprechenden Stellen des DNA-bindenden Abschnitts ein – das Ergebnis: „Verändert man diesen Enzym-Abschnitt durch Mutationen, so reichert sich KAT6A nicht mehr an CpG-Inseln an“, berichtet Liefke. „Unsere Arbeiten tragen zu einem besseren Verständnis bei, wie Veränderungen des KAT6A Enzyms zu Entwicklungsstörungen und Krebs führen können“, sagt Liefkes Mitarbeiterin Lisa Weber, die als Erstautorin des Fachaufsatzes firmiert.
Die Lebenswissenschaften gehören zu den Forschungsschwerpunkten der Philipps-Universität Marburg. Der Biochemiker Robert Liefke leitet eine Nachwuchsgruppe am Institut für Molekularbiologie und Tumorforschung der Universität. Neben seinem Team sowie weiteren Wissenschaftlerinnen und Wissenschaftlern der Philipps-Universität beteiligten sich Forscherinnen und Forscher aus München, Boston in den USA und Peking in China an der Veröffentlichung. Die Deutsche Forschungsgemeinschaft, die Fritz-Thyssen-Stiftung sowie Förderorganisationen aus China und den USA unterstützten beteiligte Forscherinnen und Forscher finanziell.
Originalveröffentlichung: Lisa Marie Weber, Yulin Jia & al.: The histone acetyltransferase KAT6A is recruited to unmethylated CpG islands via a DNA binding winged helix domain, Nucleic Acids Research 2022, DOI: https://doi.org/10.1093/nar/gkac1188