Hauptinhalt
Forschungsschwerpunkte
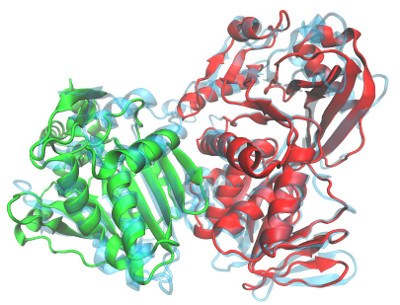
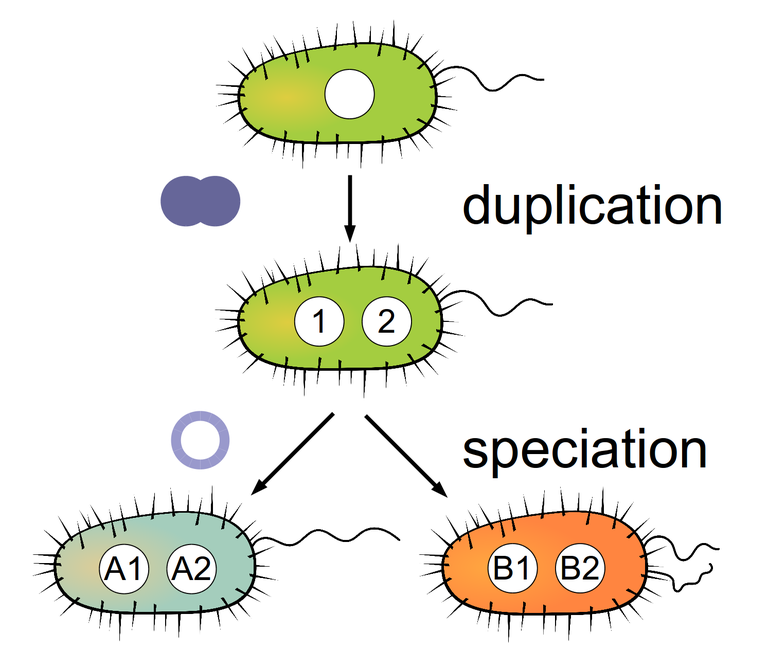
Orthologie
Orthologe Gene entstehen durch die Aufteilung einer Art zu zwei Unterarten, der sogenannten Speziation. In beiden Unterarten existiert also ein Gen gleichen Ursprungs, welches darum vermutlich auch die gleiche Funktion inne hat. Diese Information ist Grundlage für verschiedenste Fragestellungen der vergleichende Genomik. Gene welche exklusiv oder nur in einer bestimmte Variante in pathogenen Bakterien vorkommen, sind gute Ziele für die Entwicklung von Antibiotika. In der Pharmazie wird zudem oft auf Naturstoffe zurückgegriffen, deren Herstellung synthetisch kaum möglich ist. Über den Vergleich von Organismen die diesen Stoff herstellen zu anderen welche dies nicht können, lässt sich die Gruppe der relevanten Enzyme deutlich einschränken.
Dem gegenüber steht die Duplikation von Genen innerhalb einer Art. Es entstehen sogenannte Paraloge. Diese können z.B. neue Funktionen übernehmen oder sich die ursprüngliche Funktion teilen. Einen anderen Fall stellen Xenologe dar. Hierbei handelt es sich um Gene, die über horizontalen Gentransfer von anderen Organismengruppen in das Erbgut eingebracht wurden. Entsprechende Analysen sind daher auch evolutionsbiologisch von großer Bedeutung.
Die bioinformatische Kategorisierung orthologer und paraloger Gene ist sehr aufwendig, insbesondere wenn mehrere Arten miteinander verglichen werden sollen. Um diesen Vorgang zu vereinfachen und große Analysen Umsetzen zu können, haben wir unser Wissen in dem Programm Proteinortho implementiert, welches wir stetig pflegen und weiterentwickeln.
6S RNA
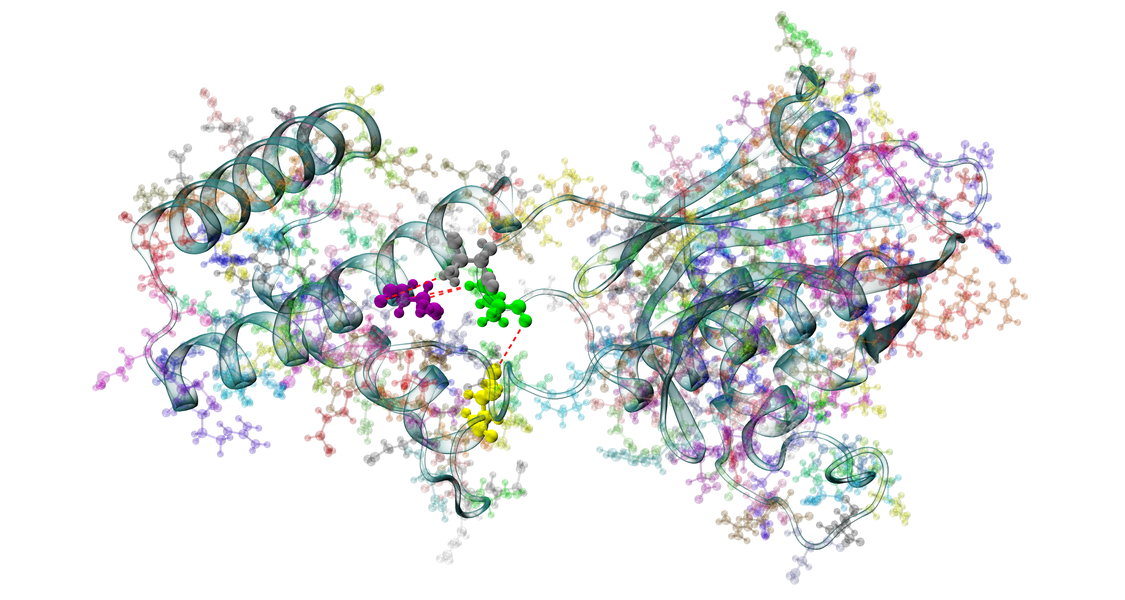
Die 6S RNA ist eine ca. 200 nt lange nicht-kodierende RNA, welche in nahezu allen Bakterien zu finden ist und in hohem Maße exprimiert wird. Obwohl seit über 50 Jahren untersucht, ist ihre genaue Funktion bis heute nicht bekannt. Wir untersuchen dieses Molekül unter verschiedenen Gesichtspunkten mit bioinformatischen als auch molekular-biologischen Methoden. Es scheint als könnte die 6S RNA ihre Funktion in den typischen Laborstämmen nicht oder nur eingeschränkt übernehmen. Diese Stämme lassen sich in der Regel gut kultivieren und transformieren, spiegeln aber nicht das genetische Setup freilebender Bakterien wider. Aus diesem Grund verwenden wir zur Funktionsaufklärung "wilde" Bakterienstämme.
Unsere bisherigen Ergebnisse legen vielfältige Einsatzmöglichkeiten im pharmazeutischen aber auch im lebensmitteltechnischen Bereich nahe. Beispielsweise könnte die Ausbreitung pathogener Keime durch gezielte Beeinflussung der 6S RNA gebremst werden. Anderseits könnte die Fermentierung von Lebensmitteln durch Manipulation der 6S RNA beschleunigt werden.
RNase P
Diese Enzym-Gruppe existiert in allem Domänen des Lebens, von Bakterien bis hin zum Menschen. Die Hauptfunktion liegt in der Prozessierung der 5'-Enden von pre-tRNAs. RNase P ist somit essentieller Bestandteil eines jeden Genpools. In der Regel handelt es sich um ein Mehrkomponenten-System aus eine RNA-Einheit und verschiedenen Proteinen. Diese unterscheiden sich z.B. von Bakterien zum Menschen deutlich, wodurch sie ein gutes pharamzeutisches Ziel darstellen. Gleichzeitig bieten die klassenspezifischen Kombinationen und Variationen von RNA und Proteinen evolutionäre Einblicke in die Abstammung der Arten aber auch horizontalen Gentransfer.
Virale Proteine
Viren haben in der Regel ein sehr kondensiertes Genom. Jedes Nukleotid hat eine Funktion. Prominente Viren wie Influenza und Ebola kodieren dabei nur für eine Hand voll Proteine. Kleine Unterschiede in der kodierenden Sequenz können dabei einen großen Unterschied in der schwere der Erkrankung oder der Wirtsspezifität eines Virus machen. Wir untersuchen diese Zusammenhänge für VP30 in aus dem Ebola-Virus und NS1 in Influenza. Ziel ist zunächst das grundlegende Verständnis der molekularen Zusammenhänge. Langfristig lässt sich die Vorhersage potentiell gefährlicher Mutation in der aktuell zirkulierenden Virenpopulation aus diesen Informationen ableiten, sodass z.B. Impfstoffe frühzeitig bereitgestellt werden können.