Hauptinhalt

Vergleichende Genomik
Sich ein einzelnes Genom anzusehen, genügt oft nicht um grundlegende Aussagen über den dazugehörigen Organismus zu treffen. Erst der Vergleich mit anderen nah und entfernt verwandten Arten erlaubt es Eigenheiten auszumachen. Mittels Art-übergreifender Analysen kann z.B. die Verbreitung pathogener Faktoren und Resistenzgenen über verschiedene Speziesgruppen oder Lebensräume hinweg betrachtet werden. Sie stellen aber auch eine gute Möglichkeit dar, um horizontalen Gentransfer zu erkennen und zu verfolgen. Mit Proteinortho arbeiten wir hierfür kontinuierlich an einem hoch-parallelen und dennoch leicht zu bedienenden Programm zur Vorhersage orthologer Gene. Darüber hinaus vergleichen wir aber auch Organismen auf genomischer Ebene, um z.B. regulatorische Unterschiede in Promotoren zu erkennen.
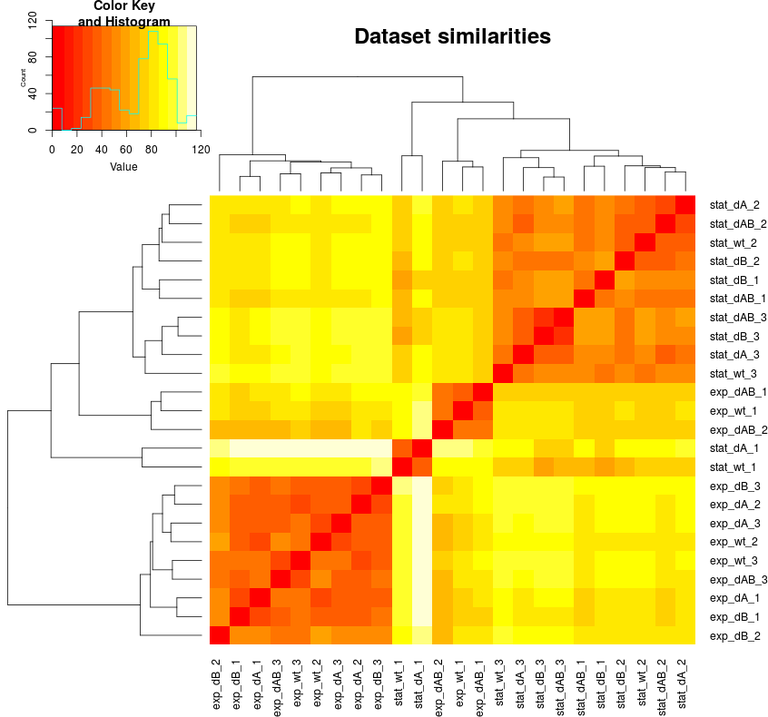
RNA-Seq
Wir befinden uns im Zeitalter des Next-Generation-Sequencing. Nahezu alle molekularbiologischen Untersuchungen generieren regelmäßig große Mengen von RNA-Seq Daten, welche je nach konkreter Fragestellung teils sehr unterschiedlich konzipiert und ausgewertet werden müssen. Unsere Analysen umfassen typischer Weise die differentielle Expression von Genen unter verschiedenen Bedingungen, die Bestimmung von Transkriptionstarts und Terminatoren, die Identifikation zirkulärer Transkripte aber auch die Prozessierung von RNAs. Über iCLIP-Experimente können darüber hinaus Protein-RNA-Interaktionen bestimmt werden. Ein Spezialgebiet stellt die Sequenzierung sehr kurzer RNAs dar (7-14 nt).
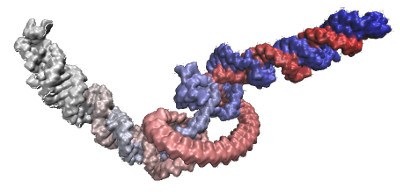
Strukturbiologie und virtuelle molekulardynamische Simulationen
3D-Modelle von Proteinen und nicht-kodierenden RNAs bieten einen detaillierten Einblick in Interaktionen auf molekularer Ebene. Interaktionsstellen, Konformationsänderungen, Strukturflexibilität und Faltungspfade können mittels molekulardynamischer Simulationen nachvollzogen und simuliert werden. Diese Simulationen erlauben in silico Vorhersagen mit denen nötige Laborexperimente effektiv eingegrenzt werden können. 3D-Simulationen auf molekularer Ebene sind extrem rechenintensiv. Dank des MaRC2 (Marburger RechenCluster 2) sind wir in der Lage Interaktionen zwischen Molekülen und deren Faltungspfade über mehrere hundert Nanosekunden zu beobachten.
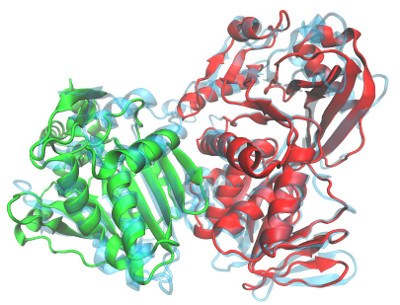
RNA Biologie und Antisense Strategien
In der Zelle transportieren RNAs nicht nur Informationen wie den Bauplan von Proteinen, sie können auch regulatorische Funktionen haben. Anders als bei Proteinen lässt sich die Funktion von RNAs in den wenigsten Fällen von ihrer Sequenz ableiten. Wir arbeiten mit der Sekundär- und Tertiärstruktur dieser Moleküle, um sie funktionell zu annotieren und gut zugängliche Bereiche für RNA-RNA oder Protein-RNA Interaktionen zu bestimmen. Diese Methoden werden eingesetzt, um z.B. regulierte Ziele kleiner RNAs zu bestimmen oder gezielt Antisense-basierte Therapien gegen bestimmte Krebsformen oder Virusinfektionen zu entwickeln. Ebenso ermöglicht ein solcher antisense-basierter Knockdown die Funktion essentieller Gene ohne genetische Manipulationen zu untersuchen.